Home > PyMOL > コマンドの基本
コマンドのように文字だけで指示を与えるとなると、とっつきにくい感じがしますが、とくに難しいことはありません。 GUIを用いたマウス操作と合わせて、コマンドを使用すると、細かい操作が楽になります。
基本的には、何を(ex:chain Aを、残基番号25~120を、グルタミン酸を・・・) どうする(ex:show 表示する、hide 非表示にする、sel 選択する、create オブジェクトを作成する、set 設定を変える・・・) という指示を与えるだけです。あとはこの基本骨格に加えてパラメータなどがついたりします。
PDBファイルを開く
fetchのあとに4桁のPDB IDを入力することでPDBファイルを開くことができます。セレクタ
何をに当たる部分です。残基番号やアミノ酸残基などを指定します。| 識別子 | 説明 | 例 | elem |
原子種 |
elem s 硫黄原子 elem o 素原子 elem fe 鉄原子 |
| resn |
残基名 |
resn cys システイン (resn glu,asp) グルタミン酸とアスパラギン酸 select (resn glu,asp and chain A) chain Aのグルタミン酸とアスパラギン酸 |
| resi |
残基番号 |
resi 20 残基番号20 (resi 83,99,105) 残基番号83と99、105 (resi 20:50) 残基番号20~50 (resi 20:50,83,105) 残基番号20~50と83、105 (resi 20:50 and chain A) chain Aの残基番号20~50 |
| chain |
Chain ID |
chain A chain A (chain A,B) chain Aとchain B |
セレクタと以下のどうするに当たるコマンドを合わせて使用します。コマンドの一部を紹介します。 より詳しい説明やその他のコマンドについてはPyMOL wiki をご参照ください。
sel 選択する
- 選択する
- 選択してオブジェクトパネルに追加する 選択するだけでなく、対象をオブジェクトパネルに任意の名前で保存します。
のようにselの後ろにスペースを空けてセレクタを入力します。例えば
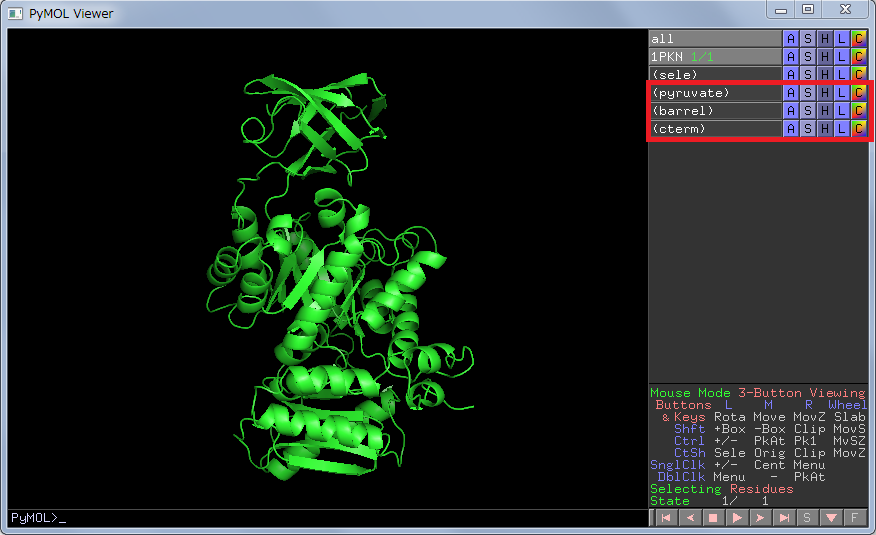
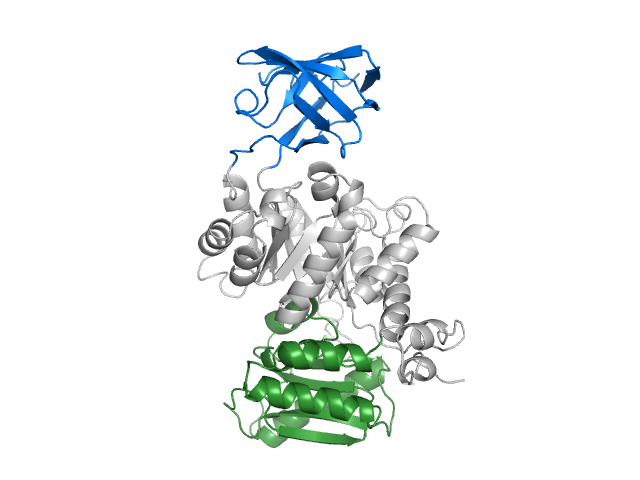
例としてマルチドメインタンパク質をドメインごとに色を変更してみます。chainごとの色分けは簡単ですが、 ドメインの場合は1つのchainなので、別々に指定する必要があります。
ピルビン酸キナーゼ(PDB ID:1PKN)は3つのドメインを持ちます。・残基番号1-116,218-388:ピルビン酸ドメイン
・残基番号117-217:βバレルドメイン
・残基番号389-530:C末端ドメイン
3つのドメインにそれぞれ適当な名前を付けて、オブジェクトパネルから色を変更します。
sel barrel, (resi 117:217)
sel cterm, (resi 389:530)
create オブジェクトを作成する
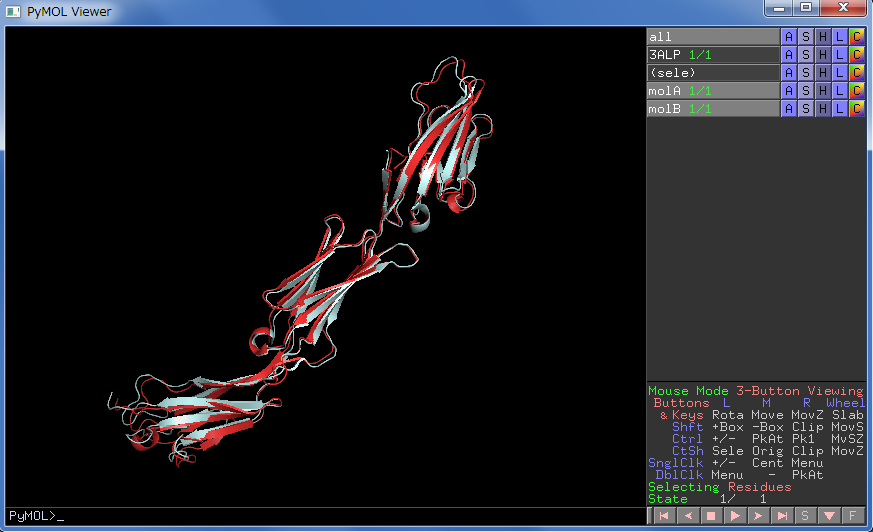
selと同じよな使い方をします。対象を複製して新しいオブジェクトとして追加します。例とし非対称単位中に存在する分子を重ね合わせてみます。PDB ID:3ALPは非対称単位中に2分子存在します。 chain AをmolA、chain BをmolBとして複製して、重ね合わせます。
create molB, chain B
align molA, molB
show 表示する
- 表示する
- 選択した対象を表示する
stickを表示する、surfaceを表示するといった感じで使用します。対象を入力していない場合はすべてが対象となります。
show surface
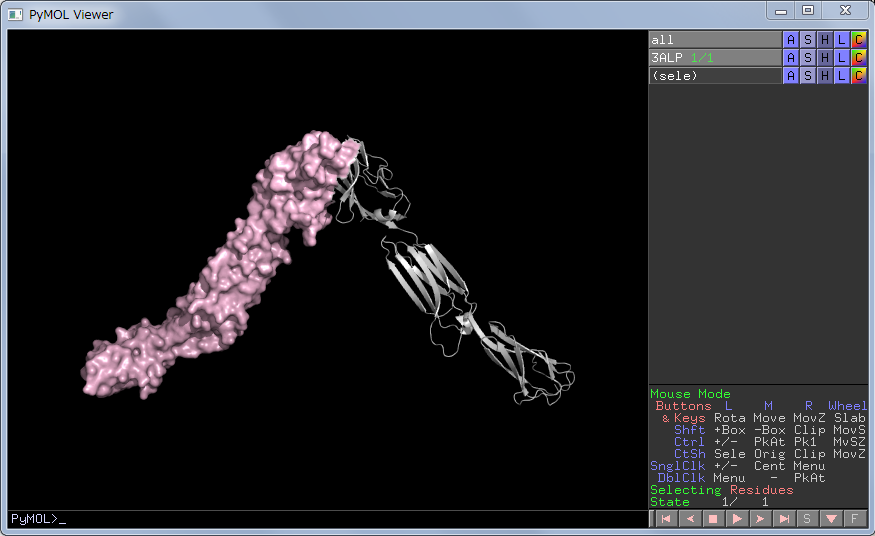
対象を入力して一部を表示することもできます。例えば、すべて非表示にしてchain Aをcartoonで、chain Bを surfaceで表示してみます。
show cartoon, chain A
show surface, chain B
set 設定を変える
setはいろんな使い方があるので説明しきれません。例として、ぱっと見できれいな図を作ってみます。
・cartoonとsurfaceの色の変更(4,5行目)
・surfaceの透過(6行目)
・背景を白に変更(7行目)
・影を消す(8行目)
show cartoon
show surface
set cartoon_color, green
set surface_color, grey70
set transparency, 0.7
bg_color white
set ray_shadow, off
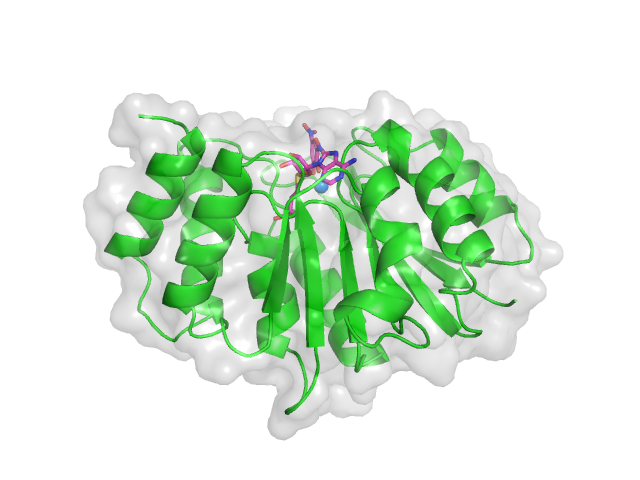
※別途リガンドと金属を表示しています。


